
Полная версия
ДНК и её человек
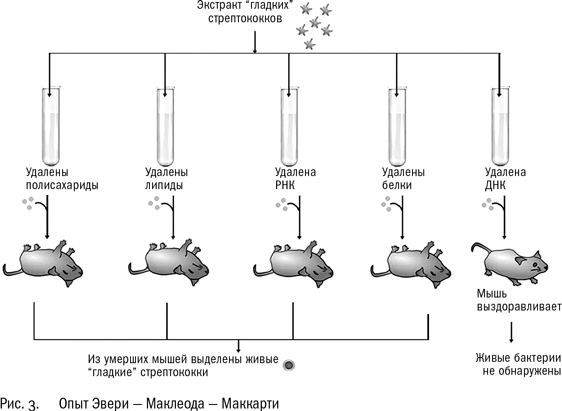
Для эксперимента Херши и Чейз выбрали бактериофаг Т 4. Бактериофаги – вирусы бактерий, одни из самых простых объектов живой природы. (Хотя насчет “живой” идут бесконечные споры. Вирусы и бактериофаги не могут размножаться вне клетки, к тому же бактериофаг можно закристаллизовать, как, например, молекулу белка. Так, может быть, их следует рассматривать как своего рода паразитические молекулярные комплексы? Не считаем ведь мы живыми прионы – белки с аномальной структурой, которые катализируют превращение нормальных клеточных белков в себе подобные и тем самым вызывают тяжелые заболевания – энцефалопатии). Так или иначе, вирусы и фаги размножаются, потомство у них похоже на родителя, и “вещество наследственности” у них должно быть. Что важно, фаг не проникает внутрь бактерии целиком: он, как шприц, впрыскивает в нее свое содержимое, оболочка остается снаружи клетки, а потом внутри бактерии образуются новые фаги.
Херши и Чейз показали, что фаги вводят в бактериальную клетку свою ДНК, а не белок, с помощью очень изящного опыта. Было известно, что белки содержат кислород, азот, углерод и серу, а нуклеиновые кислоты – кислород, азот, углерод и фосфор. Сера присутствует в белках, но не в ДНК, а фосфор – наоборот, в ДНК, но не в белке. Экспериментаторы получили две разновидности фагов: одни имели в своем составе радиоактивную серу 35S, другие – радиоактивный фосфор 32P. Иначе говоря, в одних фагах радиоактивную метку несли только белки, в других – только ДНК. Так вот, когда бактерий инфицировали фаги первого типа, метка оставалась снаружи, в растворе, а когда второго – меченый фосфор попал внутрь клетки, и новые фаги, которые вышли из этой клетки, тоже были немного радиоактивными. Вывод из этих экспериментальных данных читатель может сделать сам.
Статья Херши и Чейз вышла в 1952 г.[8] В то время уже никто не сомневался, что носителем информации должна быть именно ДНК. Было известно, какие компоненты входят в ее состав, была известна загадочная закономерность, именуемая правилом Чаргаффа: в ДНК попарно равны концентрации гуанина и цитозина, аденина и тимина. Оставалось понять, как все это организовано в пространстве, как устроена молекула. И началась большая гонка, описанная в книге Джеймса Уотсона “Двойная спираль”. Победили в ней, как всем известно, Уотсон и Фрэнсис Крик.
Помогли им в этом рентгенограммы, которые получила Розалинд Франклин – еще одна женщина, сумевшая сделать научную карьеру в эпоху, когда девушка уже могла учиться в Кембридже, но по окончании не могла быть уверена, что получит ученую степень даже при самых блестящих успехах. Рентгеноструктурный анализ – замечательный метод исследования биомолекул. Нужно получить кристалл, направить на него рентгеновское излучение и по картине дифракции лучей рассчитать распределение электронной плотности, координаты атомов, а следовательно, и структуру. Расчеты достаточно сложные, в докомпьютерную эпоху задача была, мягко говоря, нетривиальной. Неорганические кристаллы начали изучать с помощью рентгеноструктурного анализа еще в начале ХХ в. Но потом выяснилось, что кристаллы можно получить и из крупных органических молекул – это трудно, но возможно.
Поглядев на рентгенограммы Розалинд Франклин (особенно важную роль сыграла легендарная “фотография № 51”), Уотсон и Крик выдвинули предположение, что ДНК состоит из двух нитей, соединенных друг с другом азотистыми основаниями и закрученных одна вокруг другой. К 28 февраля 1953 г. Уотсон и Крик уже были уверены в своей правоте; Крик даже заявил в местном пабе, что они “раскрыли секрет жизни”. Их знаменитая статья[9] вышла 25 апреля 1953 г. (таким образом, в 2018 г. человечество отметило 65 лет знакомства с двойной спиралью). Статья заканчивалась горделиво-скромно: “От нашего внимания не ускользнуло, что специфическое взаимодействие, которое мы постулировали, сразу же предоставляет возможный механизм копирования генетического материала”. Действительно, двойная спираль не только соответствовала рентгенограмме, полученной Франклин, но и давала ответ на самый главный вопрос – каким образом информация копируется и передается по наследству!
Морис Уилкинс, получивший Нобелевскую премию 1962 г. вместе с Уотсоном и Криком, не участвовал в построении модели, но работы по изучению структуры ДНК, в том числе и работа Розалинд Франклин, были начаты во многом благодаря ему. Сама же Франклин не дожила до вручения премии – она умерла от рака в 1958 г. в возрасте 37 лет.
Теперь два абзаца биохимии, чтобы подвести итог достижениям отцов (и матерей) молекулярной биологии. Постарайтесь это пережить, а кому не хочется – просто посмотрите на рис. 4 (справа) и переходите к следующей главе. Молекула пятиатомного углевода дезоксирибозы в составе ДНК замкнута в цикл, к ней присоединены азотистое основание и фосфат. Атомы углерода в дезоксирибозе пронумерованы, от одного до пяти; цифры помечены штрихами (в отличие от углеродов азотистого основания, которые пронумерованы без штрихов). К 5’ – углероду присоединен “собственный” фосфат нуклеотида, к 3’ – углероду – фосфат другого нуклеотида, соседа по цепочке. По ним названы и концы нуклеотидной цепи – 5’ – и 3’ – конец. “Начало” цепи, ее “левый” конец (мы читаем слева направо, и последовательность нуклеотидов в ДНК нам удобнее записывать таким же образом) – это 5’ – конец. Любая цепочка ДНК (или РНК) растет от 5’ – к 3’ – концу – новый нуклеотидный остаток всегда присоединяется к 3’ – атому.
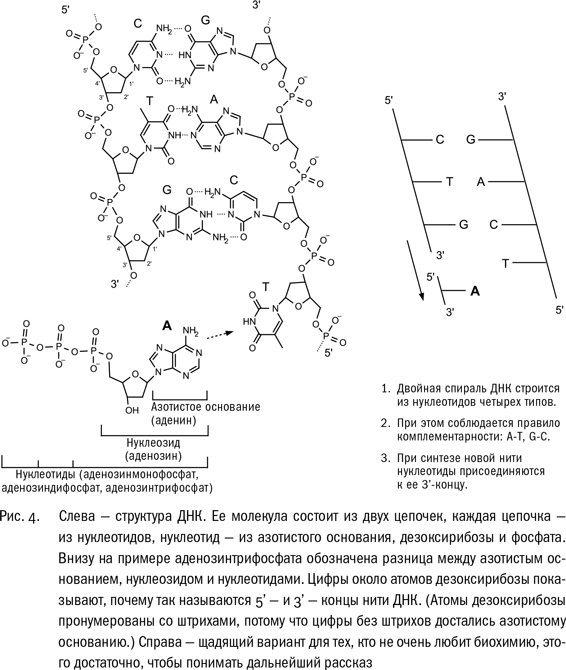
Азотистые основания – это то, благодаря чему четыре нуклеотида различаются между собой (остатки дезоксирибозы и фосфаты у всех нуклеотидов одинаковые). Два больших, аденин и гуанин (см. рис. 4), называются пуринами, а два маленьких, с одним шестичленным циклом – тимин и цитозин – пиримидинами. Это и есть те самые “буквы” А, Т, G, С, которыми записывается генетическая информация. Что существенно – этими нуклеотидами две цепочки двойной спирали держатся друг за друга. Напротив аденина всегда стоит тимин, а напротив гуанина – цитозин. Таким образом, последовательность нуклеотидов в одной цепочке однозначно определяет последовательность нуклеотидов в другой цепочке, что позволяет ДНК копироваться.
Теперь мы знаем, что такое ген!
Следующие десятилетия тоже прошли не зря. Структура молекулы – это прекрасно, однако надо было понять, каким образом ДНК копируется (реплицируется), как записанная в ней информация превращается в признаки.
Наиболее важное и удивительное свойство двойной спирали – это, конечно, заложенная в ее структуре способность к самокопированию. Если разделить две нити, то на каждой можно начать строить ее копию, в итоге получить вместо исходной двойной спирали две одинаковые и по-сестрински разделить их между сестринскими клетками. Или построить на определенном участке ДНК молекулу матричной РНК (мРНК) – инструкцию для синтеза белка. Ура, наконец-то мы узнали, что такое ген – фрагмент ДНК, в котором записана последовательность аминокислот определенного белка, плюс регуляторные участки, через которые происходит включение и выключение гена. (Правда, есть и такие гены, которые кодируют не матричную РНК и через нее белок, а просто РНК, имеющую самостоятельные функции.)
Разобрались с репликацией ДНК, расшифровали генетический код, то есть разгадали, каким образом можно записывать последовательности из 20 аминокислот четырьмя нуклеотидами. Оказалось, природа использует элегантный шифр – каждой аминокислоте соответствуют три нуклеотида; таких комбинаций существует, как нетрудно подсчитать, 64, поэтому одной аминокислоте могут соответствовать несколько триплетов – код вырожденный, как говорят математики.
Стало понятно, что ДНК в ядре клетки – это библиотека, в которой книги не выдают на дом, но позволяют снимать копии и забирать с собой. Или, в современных образах, – магазин электронных книг, который может продать бесконечное количество экземпляров той или иной книги в удобном для чтения формате. Копии книг – это матричные РНК, рибосомы (клеточные машинки для синтеза белка) читают их по триплетам и в соответствии с этими триплетами строят белок. Таким образом, поток информации идет в направлении от ДНК к РНК и затем от РНК к белку. Это и есть центральная догма молекулярной биологии, которую сформулировал Фрэнсис Крик в 1958 г.
Возможно, “догма” не самое подходящее слово. Как писал историк молекулярной биологии Хорас Джадсон в книге “Восьмой день творения” (The Eighth Day of Creation) о своем разговоре с Фрэнсисом Криком: “«Я имел в виду, что догмой называют идею, для которой нет обоснованных подтверждений. Понимаете?!» Крик издал восторженный рев. «Да я просто не знал, что значит «догма»! И мог бы с тем же успехом назвать ее Центральной Гипотезой или как-то в этом роде. Это я и хотел сказать. Догма – только слово-крючок»”. В любом случае сегодня центральная догма “ДНК → РНК → белок” ни у кого не вызывает ни малейших сомнений: обоснованных подтверждений более чем достаточно. Хотя теперь известно, что информация может передаваться от РНК к ДНК (например, в жизненном цикле некоторых вирусов с РНК-геномом), общей картины это не меняет. Магистральный поток информации направлен от ДНК к белкам – строителям и строительным материалам всего живого.
Как читать ДНК
Фредерик Сенгер и его метод секвенирования
Конечно, все захотели читать ДНК – черпать информацию о жизни прямо из источника. Но как читать буквы, если эти буквы – молекулы?
Необходим был удобный метод определения нуклеотидной последовательности, и такие методы стали появляться. Правда, большая часть их сегодня имеет лишь историческую значимость: для нынешних биологов “плюс-минус” секвенирование или “секвенирование по Максаму – Гилберту методом химической деградации” – что-то вроде микроскопа Левенгука.
Слово “секвенирование”, собственно, и означает “определение последовательности” (от англ. sequence); говорят о секвенировании ДНК, РНК, белков. Предложенное в 1970 г. секвенирование по Максаму – Гилберту, если коротко, подразумевало расщепление ДНК в растворах, организованное таким образом, чтобы получались молекулы всех возможных длин. Но это не самый рациональный подход. ДНК – именно та молекула, которая умеет копироваться сама на себе. Если взять у клетки ферменты, которые работают с ДНК, и научиться их использовать в наших целях, можно добиться многого. Почему бы, например, вместо того чтобы нарезать ДНК столькими способами, сколько в ней букв, не нарастить на ней дочерние цепи всех возможных длин? На этой идее основано секвенирование по Сенгеру – метод, также изобретенный в 70-е гг. прошлого века и благополучно доживший до наших дней.
Английский биохимик Фредерик Сенгер (1918–2013) – один из четырех человек, получивших две Нобелевские премии, и единственный, у которого обе – по химии (1958 и 1980 гг.): за определение структуры белка инсулина и за метод секвенирования ДНК. В 1975 г. Сенгер в совместной статье с Аланом Коулсоном представил метод “плюс-минус” секвенирования[10]. С помощью этого метода группа Сенгера почти полностью прочла геном бактериофага φX174 (5386 нуклеотидов) – по тем временам большой успех[11]. Однако все эти достижения затмило секвенирование по Сенгеру методом терминаторов, он же метод обрыва цепи, или дидезоксиметод[12]. Но сначала нужно объяснить, как молекулы ДНК сортируют по размеру с помощью электрофореза.
Мы помним, что ДНК – это кислота. Кислотные свойства определяются остатками фосфорной кислоты в ее составе – фосфатами. Из школьного курса известно, что анионы кислоты заряжены отрицательно: H3PO4 → H+ + H2PO4– . Поэтому, если через раствор, содержащий ДНК, пропустить ток, молекулы ДНК направятся к положительно заряженному электроду. А если раствор заменить гелем – несъедобным желе, молекулярной сеткой, заполненной жидкостью? Тогда молекулы ДНК не поплывут к плюсу туманным облачком, а каждая станет продвигаться со своей скоростью – чем длиннее молекула, тем труднее ей будет просачиваться через ячейки геля. Если же сделать в геле углубления (лунки) у отрицательного электрода, поместить в них ДНК и включить ток, от минуса к плюсу протянутся дорожки, и в них будут полоски, в каждой – молекулы ДНК определенного размера. Это называется “электрофорез ДНК”, или просто форез.
Теперь можем рассказать, как работает метод Сенгера. Реакционную смесь делят на четыре части. В каждую из четырех добавляют праймер (затравку для синтеза) – короткую молекулу ДНК, комплементарную началу участка, который нужно секвенировать. Праймер связывается с этим участком, образуя с ним двойную спираль. (Исследуемую ДНК перед этим, конечно, надо “расплести”, сделать однонитевой.) Фермент ДНК-полимераза, используя анализируемую ДНК в качестве матрицы, начинает наращивать праймер, соединяя в цепочку нуклеотиды. К обычным нуклеотидам в реакционной смеси добавлены необычные. Во-первых, некоторые нуклеотиды содержат радиоактивную метку (потом объясню зачем). Во-вторых, в каждой из четырех смесей небольшое количество одного из четырех нуклеотидов модифицировано – лишено OH-группы. К такому нуклеотиду (дидезоксинуклеотиду) нельзя присоединить следующий. А количество подобрано таким образом, чтобы среди новых цепочек были оборванные на каждом аденине – в одной пробирке, тимине – в другой, гуанине – в третьей, на каждом цитозине – в четвертой. И если потом внести реакционные смеси в лунки и провести электрофорез, получатся “лесенки”. Сложно, но станет яснее, если посмотреть на рис. 5 и 6.
Автоматическое распознавание
Как это происходило на практике? О-о… Классическое секвенирование по Сенгеру – процедура, которая у старшего поколения ассоциируется с золотым веком молекулярной биологии, когда было мало простого, покупного и готового и все умели работать руками, не то что сейчас. Не удержусь и расскажу подробнее: я ее еще застала.
Итак, гель нам понадобится не агарозный, как для крупных фрагментов, сильно различающихся по размеру, а полиакриламидный и очень тонкий. Разделить молекулы ДНК, различающиеся всего на один нуклеотид, – серьезная задача. Гель готовим из акриламида (канцероген; распишитесь, студенты, что поняли, а также насчет радиоактивной метки – хоть фосфор-32 и смешной по активности изотоп, но все-таки пить его не надо). Горячую прозрачную жидкость заливаем в пространство между двумя идеально плоскими стеклами размером примерно А3 с зажатыми по краям полосками пластика – спейсерами. Стеклянная струйка сбегает вниз, гель заливается, заливается, залива… черт-черт, пузырь застрял и не всплывает! Подхватываем что-нибудь твердое, вроде ножниц, стучим по стеклу деликатно, но сильно: уходи, пузырь, уходи! Если повезло, пузырь неохотно поднимается вверх, если нет – застывает в геле, делая значительную его часть непригодной для фореза, а студент слышит у себя за спиной: “Ничего, ручки кривые, зато старательный”… Сверху заливаем чуть менее крепкий гель, в который вставляют гребенку, чтобы получились лунки. Ждем. Гель застыл. В реакционные смеси добавляем глицерина, чтобы смесь не всплывала в растворе, а сиропчиком оседала на дно лунки, а также синего и фиолетового красителя с отрицательно заряженными молекулами, чтобы видно было, достаточно ли далеко прошел форез: раствор ДНК сам по себе прозрачен, по нему не поймешь. Вносим смесь в лунки. Аккуратненько, только не мимо лунки, а то будет каша. Не путаем, что куда, запоминаем, а лучше записываем. Готово. Ставим электрофорез. На табло источника питания четырехзначное число, обозначающее вольты, кстати, распишитесь, студенты, что поняли насчет высокого напряжения.
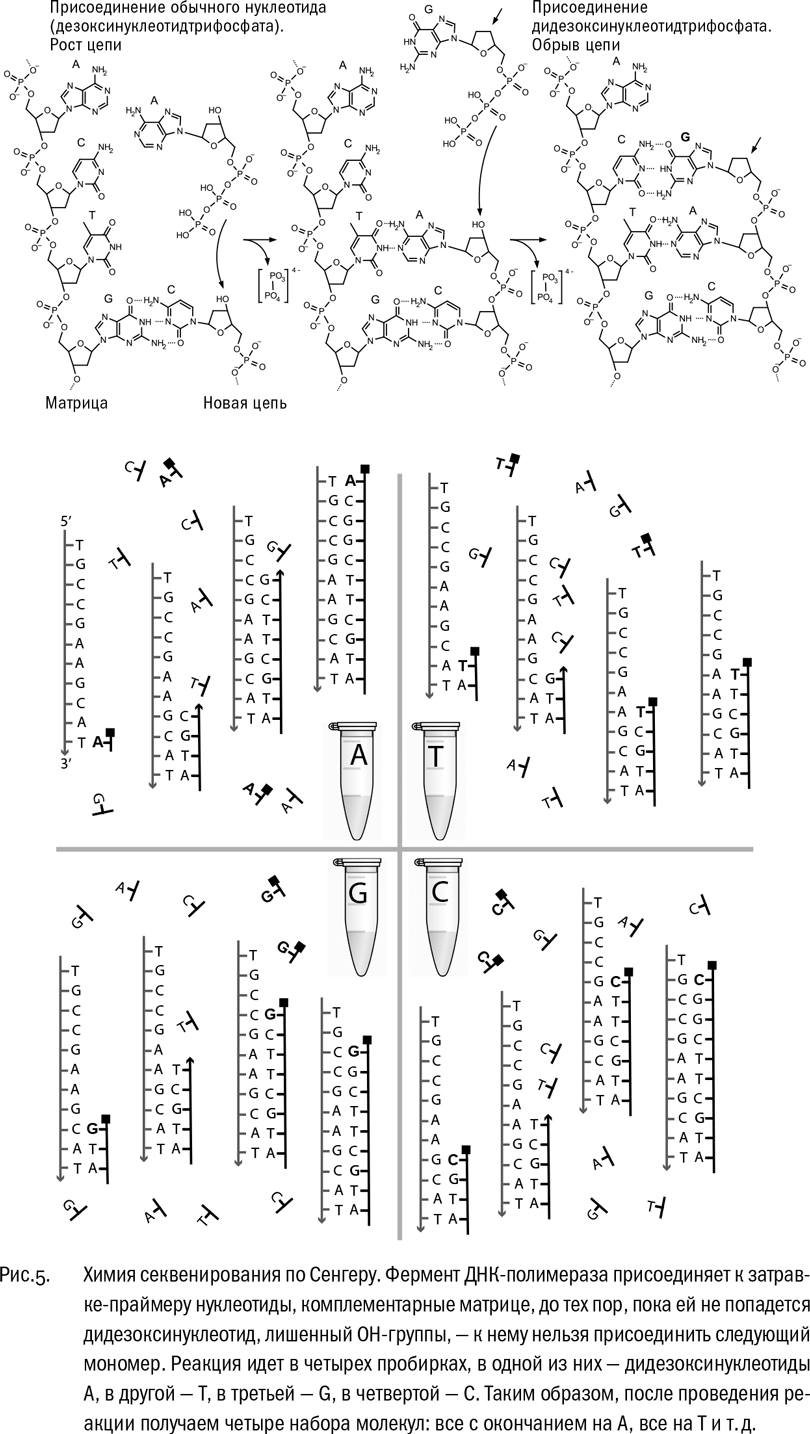

По прошествии изрядного времени, когда мы видим, что синее и фиолетовое пятна проехали к плюсу сколько надо, снимаем гель и сушим. Он у нас слегка радиоактивный, мы не забыли? При синтезе меченые нуклеотиды включались в цепочки ДНК, поэтому все новые молекулы фонят. Мы берем гель в темную комнату и аккуратно прижимаем его к рентгеновской пленке размером с наше стекло. Зажимаем в металлическую коробку и оставляем, скажем, до завтра. Потом проявляем пленку – и вуаля: если все сделали правильно, на прозрачной пленке темнеют полосочки, выстроенные лесенкой. Это называется “радиоавтограф геля”. Каждая полоска соответствует нуклеотиду ДНК. Кстати, сама идея метить молекулы ДНК, заставляя полимеразу включать в них нуклеотиды с радиоактивными изотопами, для удобства последующих наблюдений тоже принадлежит Фредерику Сенгеру.
Вот теперь наконец-то читаем нашу ДНК! Сначала две полоски на левой дорожке, затем одна на правой, затем на второй слева – ААGТ… Одному это расшифровывать не с руки. Зовешь помощника, даешь ему в руки линейку, велишь диктовать, а сам вбиваешь в компьютер буквы ДНК – текст, который никто еще не читал, кроме вас двоих и Господа Бога, если он вникал в такие мелочи, а не предоставил все эволюции. За один раз на четырех дорожках можно прочесть несколько сотен нуклеотидов, в идеале до тысячи. (Для сравнения, “плюс-минус” секвенирование давало около 80 нуклеотидов.) Уф-ф.
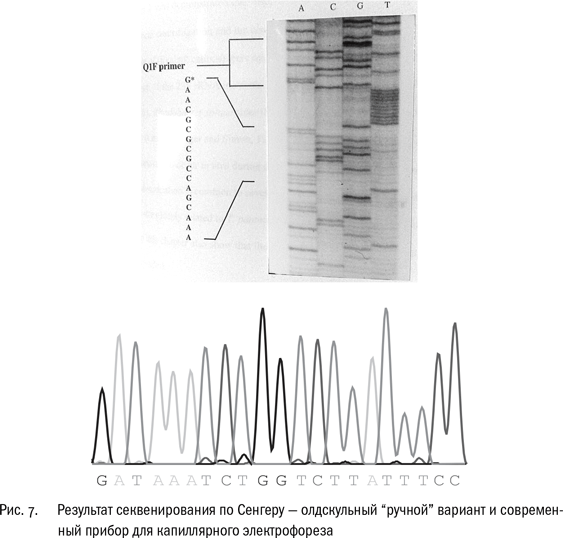
Теперь, с появлением приборов-секвенаторов, взаимодействие человека и ДНК стало менее интимным и утомительным. Человек ставит реакционную смесь в прибор и идет пить кофе… то есть писать обзор литературы для статьи. Никакой романтики преодоления трудностей. (Шучу. На самом деле трудности теперь в других местах – например, там, где начинается обработка огромного количества данных.)
Принцип метода остается тем же, что и в классическом секвенировании по Сенгеру, – синтез четырех наборов нуклеотидных цепочек, кончающихся на А, на Т, на G и на С. Только электрофорез теперь происходит не в плоском геле, а в капилляре, из которого синтезированные молекулы выходят поочередно, от самых коротких к самым длинным. И метка не радиоактивная, а флуоресцентная: каждый терминаторный нуклеотид светится своим цветом, условно говоря, А – зеленым, Т – красным, С – синим, G – желтым. (Реакционных смесей уже не четыре, а одна!) Регистрирующее устройство фиксирует вспышки на выходе из капилляра и отмечает пики свечения каждого цвета. Прибор выдает график с четырьмя кривыми, где пики соответствуют нуклеотидам; последовательность нуклеотидов сохраняется в памяти компьютера.
Первые автоматические секвенаторы начала поставлять фирма Applied Biosystems (1986). Они использовали принцип, разработанный в Калифорнийском технологическом институте, в лаборатории Лероя Худа. Что интересно, в первоначальном варианте секвенирования от Applied Biosystems реакционных смесей было четыре, и флуоресцентную метку несли не дидезоксинуклеотиды, а праймеры. Это было своего рода промежуточное звено между сенгеровским методом и последующим автоматизированным – реакция идет в четырех смесях, но все продукты бегут по одной дорожке электрофореза. Эволюция техники иногда похожа на эволюцию живых существ: полезные изменения накапливаются последовательно.
Applied Biosystems (к тому моменту подразделение компании PerkinElmer) принимала непосредственное участие в создании компании Celera Genomics, основателем которой был знаменитый Крейг Вентер, человек, который многое сделал для того, чтобы чтение ДНК вышло на новый уровень – от сотен и тысяч нуклеотидов к целым геномам. Celera Genomics вскоре прославилась как главный конкурент международного проекта “Геном человека”, а Крейг Вентер, как он сам пишет в своей автобиографии, был одним из первых клиентов Applied Biosystems еще в то время, когда руководил лабораторией в Национальных институтах здравоохранения. Важную роль в его последующих успехах, да и вообще в секвенировании генома человека сыграли автоматические секвенаторы. И наоборот: поставленная грандиозная задача – 3 млрд нуклеотидов, во времена, когда и тысячи считались успехом! – способствовала автоматизации секвенирования.
В 1995 г. Институт геномных исследований Крейга Вентера (TIGR) прочитал первый полный геном бактерии Haemophilus influenzae (1,8 млн нуклеотидных пар). И заодно, “просто чтобы проверить метод”, геном Mycoplasma genitalium (0,58 млн н.п.) – той самой бактерии, на основе которой Крейг Вентер с соавторами в первом десятилетии будущего века начнет создавать синтетический геном. Секвенирование полных бактериальных геномов микробиологи восприняли как сенсацию, историческое событие, Вентеру на конференции, когда он объявил об этом, аплодировали стоя. В 1998 г. был секвенирован геном многоклеточного организма – круглого червя Caenorhabditis elegans (100 млн н.п.).
Проект “Геном человека” стартовал в 1990 г. О получении первой “черновой” последовательности руководитель международного проекта Фрэнсис Коллинз и Крейг Вентер торжественно объявили 26 июня 2000 г. в Белом доме. Окончательное завершение проекта было анонсировано в апреле 2003 г.
Кстати: многие издания писали тогда, что, мол, “расшифрован генетический код человека”. Некоторых биологов это бесило почти так же, как “ваше ДНК”. Дело в том, что по-русски кодом принято называть шифр – правило соответствия между двумя системами символов, в нашем случае – между аминокислотами белка и нуклеотидными триплетами. Генетический код, то есть соответствие аминокислот и триплетов, у человека тот же, что у всех живых организмов, и расшифрован он давно! По-английски же кодом можно назвать и шифр, и шифровку, так что аналогичный английский заголовок не кажется глупым. Впрочем, сейчас уже и в русском языке так прочно прижился “код” как текст компьютерной программы, что это значение задним числом легитимизирует и “генетический код человека”.
Стоимость проекта “Геном человека” составила $3 млрд – по доллару за букву. Сейчас цена вопроса – порядка $1000 геном (в России пока подороже), ближайшая цель конкурирующих фирм – снижение до сотен долларов.
Во всем этом Сенгер уже не принимал непосредственного участия. В 1983 г. он ушел в отставку и прожил три десятилетия, с удовольствием работая в своем саду. Младшие коллеги о нем не забывали – тот же Крейг Вентер с гордостью приводит факсимиле поздравительной записки от Сенгера по поводу расшифровки генома H. influenzae. В 1992 г. в Великобритании был создан Институт Сенгера – некоммерческий геномный исследовательский центр. Двукратный нобелиат напутствовал коллег словами: “Пусть только попробуют не добиться успеха”. Но сам он не любил публичности и даже отказался от рыцарского звания за научные заслуги. Умер Фредерик Сенгер в 2013 г. в возрасте 95 лет. Вот как он объяснял, почему так рано удалился от дел: “Я и сам не думал об отставке, пока внезапно не осознал, что через несколько лет мне будет 65 и я буду иметь право перестать работать и заняться чем-то, чего я всегда хотел и на что не имел времени. Это возможность выглядела неожиданно привлекательной, особенно потому, что наша работа достигла высшей точки с методом ДНК-секвенирования, и я в некотором роде чувствовал, что продолжать – значит двигаться к низшей точке. Решение, что я принял, было мудрым – не только потому, что я получил огромное удовольствие от своего нового образа жизни, но и потому, что старение не улучшило мою производительность в лаборатории, и я думаю, что если б я продолжил работать, то мог бы найти это разочаровывающим и чувствовал бы вину за то, что занимаю место, нужное молодым людям”[13].
Новое поколение выбирает…
В современных научных статьях по исследованию ДНК часто можно встретить аббревиатуру NGS. Это расшифровывается как next generation sequencing, методы секвенирования нового поколения – собирательное название для новейших методов, не использующих cенгеровскую терминацию. Все они появились после двухтысячного года, все требуют довольно сложного оборудования и программного обеспечения. Для большинства из них ДНК надо сначала фрагментировать – порезать на фрагменты в несколько сотен нуклеотидов, а затем состыковывать прочтенные кусочки текста в единую последовательность. Часто NGS называют также “высокопроизводительным”, или “параллельным”, секвенированием, потому что одновременно читается множество кусочков ДНК.
Подробно про каждый метод рассказывать не будем, только общий принцип в двух словах.
Конец ознакомительного фрагмента.
Текст предоставлен ООО «ЛитРес».
Прочитайте эту книгу целиком, купив полную легальную версию на ЛитРес.
Безопасно оплатить книгу можно банковской картой Visa, MasterCard, Maestro, со счета мобильного телефона, с платежного терминала, в салоне МТС или Связной, через PayPal, WebMoney, Яндекс.Деньги, QIWI Кошелек, бонусными картами или другим удобным Вам способом.


